Erbgut als Zollstock für die Nanowelt
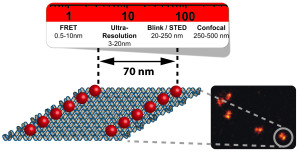
„Ähnlich wie bei den Markierungen auf einem gewöhnlichen Lineal dienen hier Punkte mit einer bestimmten Anzahl fluoreszierender Farbstoffmoleküle als Markierung“, berichtet Philip Tinnefeld, Leiter der Forschungsgruppe an der Technischen Universität Braunschweig. Unterlage für die Markierung sind lange DNA-Moleküle, die mithilfe vieler kurzer DNA-Abschnitte gefaltet werden wie eine Origami-Struktur. Je nach Bedarf können so unterschiedliche – genau bekannte – Abstände eingestellt werden, weil die Abmessungen und der Aufbau der DNA-Moleküle bestens bekannt sind. Auf den Faltstrukturen sind dann im definierten Nanometer-Abstand einzelne Fluoreszenz-Moleküle angebracht, die die Markierungen bilden. Optimal eingestellte superauflösende Mikroskope können diese Punkte dann scharf darstellen.
Mit herkömmlichen Lichtmikroskopen müssten Punkte kleiner als rund 200 Nanometer, also Millionstel Millimeter, eigentlich unscharf bleiben. Die sogenannte "Abbe'sche Beugungsgrenze" besagt, dass hier – wegen der Wellenlängen des sichtbaren Lichts – die Auflösung eines normalen Mikroskops seine Schärfegrenze erreicht. Elektronenmikroskope hingegen, die wesentlich kleinere Strukturen scharf zeigen, eignen sich nicht für das Beobachten lebender, aktiver Zellen, weil hier die Probe im Vakuum liegt und mit harten Elektronenstrahlen beschossen wird. Durch die verschiedenen mittlerweile entwickelten Superauflösungstechniken hingegen ist für Lichtmikroskope heute auch der Bereich zwischen 6 und 200 Nanometer zugänglich. Hier findet die Braunschweiger Entwicklung ihren Einsatz, um die verschiedenen Methoden – wie FRET, Ultra-Resolution, Blink und STED – und auch einzelne Geräte miteinander zu vergleichen.
Der Vorteil der neuen Nanometer-Maßstäbe ist, dass sie sich identisch auch in großer Zahl herstellen und je nach Messaufgabe quasi umprogrammieren lassen. Auch stehen verschiedene Fluoreszenz-Moleküle stehen als Leuchtpunkte zur Verfügung. Tinnefeld und Kollegen haben erste Schritte mit der Methode bereits vor drei Jahren erarbeitet.
Physiker machen chemische Bindungen sichtbar
Waage für einzelne Biomoleküle
Supermikroskop entreißt Schwarzem Tod ein Geheimnis
Biotreibstoff: Bis auf Nanometer genau in die Blätter von Energiepflanzen schauen
Scharfe Mikroskopbilder ohne Linsen
Farbiger 3D-Blick in die Zelle

 Was Sie schon immer über Mikroben wissen wollten
Was Sie schon immer über Mikroben wissen wollten Energiewende: Schluss mit Schwarz-Weiß-Strategien
Energiewende: Schluss mit Schwarz-Weiß-Strategien Nicole Bender und Lotte Habermann-Horstmeier: Evolution und Gesundheit
Nicole Bender und Lotte Habermann-Horstmeier: Evolution und Gesundheit 