Supermikroskop filmt Prozesse in lebenden Zellen
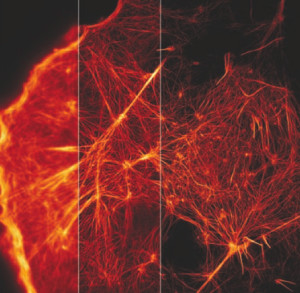
Am Howard Hughes Medical Institute in Chevy Chase optimierten Eric Betzig und seine Kollegen ihr Fluoreszenz-Verfahren, um Prozesse in lebenden Zellen über einen längeren Zeitraum filmen zu können. Der Vorteil dieser SIM-Mikroskopie (Structured Illumination Microscopy) war dabei die geringe Lichtmenge, um die Fluoreszenz-Marker in den Zellen zum Leuchten anzuregen. So wurden die Zellen weniger unter Stress gesetzt und hielten nach Betzigs Aussage einer Beobachtung länger stand als mit dem hochauflösenden Fluoreszenz-Verfahren der sogenannten STED-Mikroskopie (Stimulated Emission Depletion), die Stefan Hell entwickelt hatte.
„Schalte einfach nicht alle fluoreszierenden Moleküle an“, beschreibt Betzig den Kern seiner Methode. So aktivierte er nicht alle Fluoreszenzmarker mit einem flächendeckenden Lichtkegel, sondern beleuchtete mit einem strukturierten Lichtmuster nur einzelne Bereiche der Zelle. Darauf sendeten die Marker-Moleküle ihr Fluoreszenzlicht aus, das mit einem lichtempfindlichen Detektor aufgezeichnet werden konnte. Mit einem zweiten Lichtpuls deaktvierte er die fluoreszierenden Moleküle wieder. Beide Vorgänge lieferten erste Daten, um daraus im Computer Bilder mit einer räumlichen Auflösung von bis zu 62 Nanometern berechnen zu können.
Für die Filmaufnahmen gelang es Betzig und Kollegen nun, diese partielle Aktivierung und Deaktivierung der Marker-Moleküle 25 Mal in einer Drittel Sekunde durchzuführen. Die Menge der Daten reichte aus, um dynamische Prozesse auch mit hoher zeitlicher Auflösung aufzeichnen zu können. So entstanden kurze Videos, die die Wanderung von Proteinen durch eine Zellmembran oder die Bewegungen von Ausbuchtungen auf der Zelloberfläche, den Caveolen, zeigten.
Beide Methoden bieten für die biologische Forschung Vorteile. Die STED-Mikroskopie von Stefan Hell erreicht eine sehr hohe räumliche Auflösung von bereits wenigen Nanometern. Das SIM-Verfahren von Eric Betzig liefert zwar nicht so detaillierte Bilder, erlaubt aber eine längere Beobachtung dynamischer Prozesse, da die Zelle weniger geschädigt wird. Um Biologen das Potenzial des SIM-Verfahrens nahe zu bringen, lädt Betzig interessierte Forscher derzeit ein, ihre Zellproben an seinem Institut zu untersuchen.

 Was Sie schon immer über Mikroben wissen wollten
Was Sie schon immer über Mikroben wissen wollten Energiewende: Schluss mit Schwarz-Weiß-Strategien
Energiewende: Schluss mit Schwarz-Weiß-Strategien Nicole Bender und Lotte Habermann-Horstmeier: Evolution und Gesundheit
Nicole Bender und Lotte Habermann-Horstmeier: Evolution und Gesundheit 